
어영연세대학교 원주의대 원주세브란스 기독병원
진단검사 결과의 보고에 실제 인공지능을 적용한 경험을 토대로 기본적인 개념과 발전 방향에 대하여 간략히 살펴보았다.
1. 전통적인 기계학습과 딥러닝 인공지능은 웹 검색, 소셜 네트워크에서의 콘텐츠 필터링, 전자상거래 웹사이트 등과 같은 현대 사회의 다양한 측면에 이용되고 있다. 인공지능의 대표적인 하위 분야인 기계학습은 지도학습, 비지도 학습의 2가지 형태로 의학 전분야에 이용되고 있다. 선형 회귀분석, 로지스틱 회귀분석은 의학에서 보편적으로 사용되고 있는 지도학습 알고리즘이다. 선형 회귀분석, 로지스틱 회귀분석, 그리고 콕스 비례위험 모형은 공통적으로 한 층의 선형식만을 갖고 있어서, 비선형적인 성질을 갖는 복잡한 데이터를 표현하기에는 한계가 있다. 그래서 상기 언급된 모델들은 shallow classifier라는 별칭을 갖기도 한다[1]. 하지만, 의학에서 보편적으로 사용되고 있는 선형 회귀분석, 로지스틱 회귀분석의 중요한 장점으로는 학습된 모델에 대한 해석력(explainability)이 높다는 것이다. 예를 들면, 저밀도지단백 콜레스테롤을 예측하는 모델에서 고밀도지단백 콜레스테롤에 해당되는 파라미터는 – 1이다. 이는 저밀도지단백 콜레스테롤은 고밀도지단백 콜레스테롤과 음의 상관성을 갖고 있음을 직관적이면서도 정확하게 파악할 수 있다. 반면에 딥러닝은 독립변수라 불리는 입력값 (input variable, predictor)들에 대해 수 십 개에서 수 천 개에 달하는 파라미터를 갖고 있다. 따라서 입력 값과 예측값 사이의 1:1 관계성에 대한 해석력이 현저히 떨어지며, 입력 변수의 차원 변화와 그들 사이의 영향력을 고려한다면, 설명력은 더욱더 낮아지게 된다. 많은 연구자들은 이러한 딥러닝에 블랙박스라는 별칭을 붙이게 되었다. 하지만, 이러한 딥러닝은 실제 의학 현장에서 나오는 복잡한 데이터의 특성을 보다 정확하고 세밀하게 나타낼 수 있다.
그러면 우리는 어떤 기계학습방법을 사용해서, 예측 모델을 개발할 것인가? 한국에는 “천 리 길도 한 걸음부터”라는 속담이 있다. 모든 일에 기초부터 차근차근 진행할 때, 큰 일을 해낼 수 있다는 것을 비유하는 표현으로 예측모델 구축에도 이 속담을 적용할 수 있다. 해석력이 좋은 shallow classifier로 예측모델을 구축하고, 동일한 데이터 혹은 데이터 구조에 차근 차근 복잡한 기계학습 및 딥러닝을 적용하고, 성능의 향상 정도를 관찰 및 비교 분석을 하면 된다[2]. 예를 들면, 저밀도지단백 콜레스테롤을 예측하기 위해 많은 선형 회귀모델이 개발되었다[3]. 이후, 클러스터링 기법[4], 차원 확대(커널 기법)[5] 등이 저밀도지단백 콜레스테롤 예측모델 구축을 위해 사용되었다. 이를 바탕으로 원주세브란스기독병원 진단검사의학과는 딥러닝을 이용하여 저밀도지단백 콜레스테롤 예측모델을 구축하였다[6]. 전통적인 기계학습을 통해 충분한 해석력을 확보하고(Fig 1), 딥러닝을 통해 복잡한 실제 데이터에 대한 향상된 정확도를 얻는 과정이 바람직하다(Fig 2).
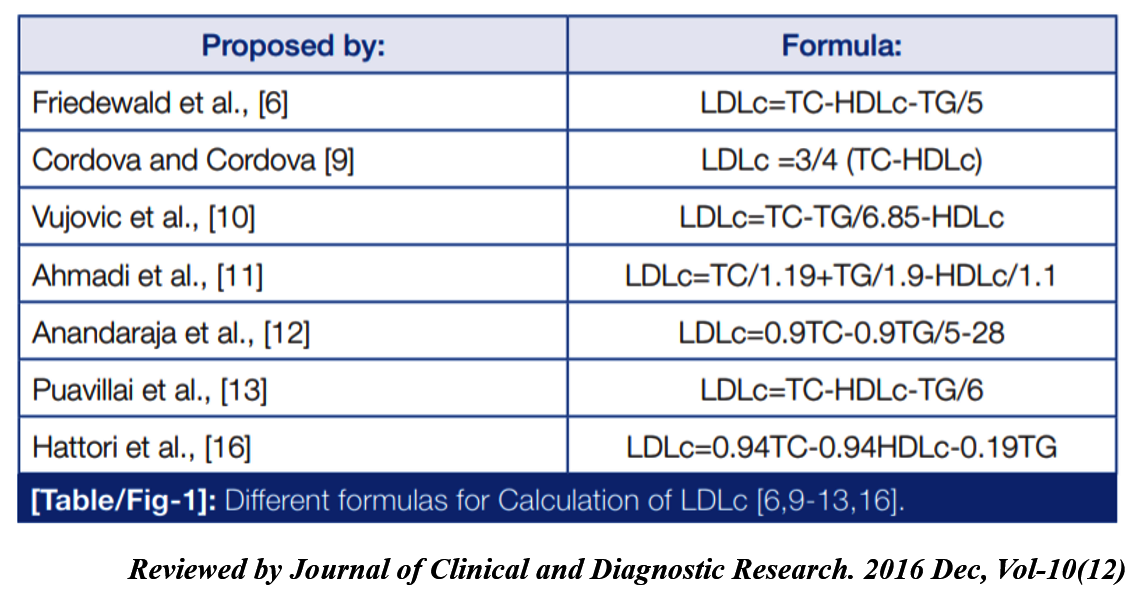
Fig 1
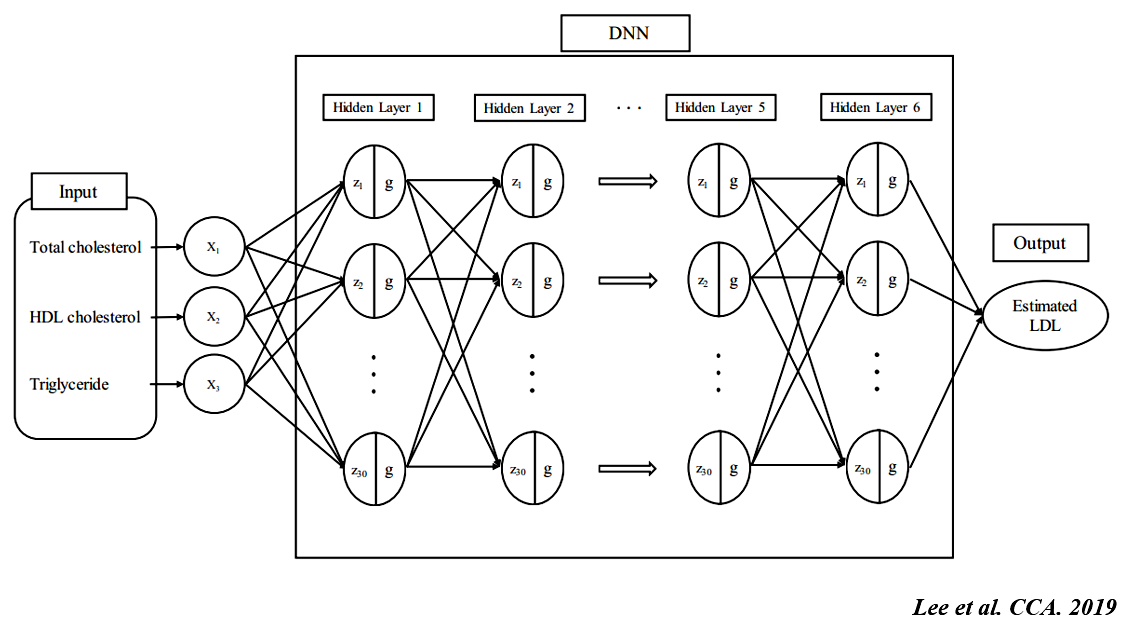
Fig 2
2. 병원 데이터베이스에 딥러닝 적용 의료의 질 향상과 의료비 감소는 의학 분야에서 인공 지능의 적용의 중요한 목적들이다. 쉽게 말하자면, 적은 비용으로 양질의 의료를 할 수 있는 환경을 만드는 것이 인공지능의 중요한 역할이라 할 수 있다. 고지혈증 약은 전세계적으로 가장 많이 처방되는 약제 중 하나이고, 약제의 효과를 파악하는 데 중요한 임상 마커로 저밀도지단백 콜레스테롤이 있다. 하지만 저밀도지단백 콜레스테롤은 추가적인 비용이 지불되어야 측정이 가능하고, 측정 방법도 다양하다. 그래서, 전세계적으로 저밀도지단백 콜레스테롤을 직접 측정하기보다는 1970년대 구축된 예측모델을 통해서 추정값을 사용해 왔다[3]. 최근 콜레스테롤 추정값 산출을 위한 딥러닝 모델을 실제 병원 데이터 베이스에 성공적으로 적용을 하였다[7]. 이 과정에 서 두 가지 딥러닝의 장점을 발견하였다. 첫 번째는 딥러닝의 적용은 기존 병원데이터베이스 플랫폼의 수정이 크게 필요하지 않다. 즉, 딥러닝의 최종 산물은 간단한 메트리스 형태의 파라미터이기 때문에 병원 데이터베이스의 연동을 위한 수정은 거의 필요하지 않다(Fig 3). 두 번째는 기존의 딥러닝 모델을 상향된 성능의 모델로 교체하는 것이 용이하다는 점이다. 전통적인 기계학습 대부분은 데이터가 추가될 때, 이전에 학습된 모델을 사용할 수 없고, 처음부터 모델 학습을 시작한다. 하지만, 딥러닝은 기존의 개발된 모델의 파라미터를 새로운 데이터를 통해서 파인 튜닝 하는 것이 가능하고 이를 전이학습이라고 한다[7] (Fig 4). 진단검사의학과에서 딥러닝 모델을 적용 하여 실시간으로 의료진에게 보고하기 위해서는 딥러닝 모델의 구축과 함께 딥러닝을 시행하는(계산을 수천 번 이상 반복하는) 서버가 필요하다. 그러므로 딥러닝을 이용하여 결과 보고를 하는 경우에는 급여 수가 반영이 요구된다.
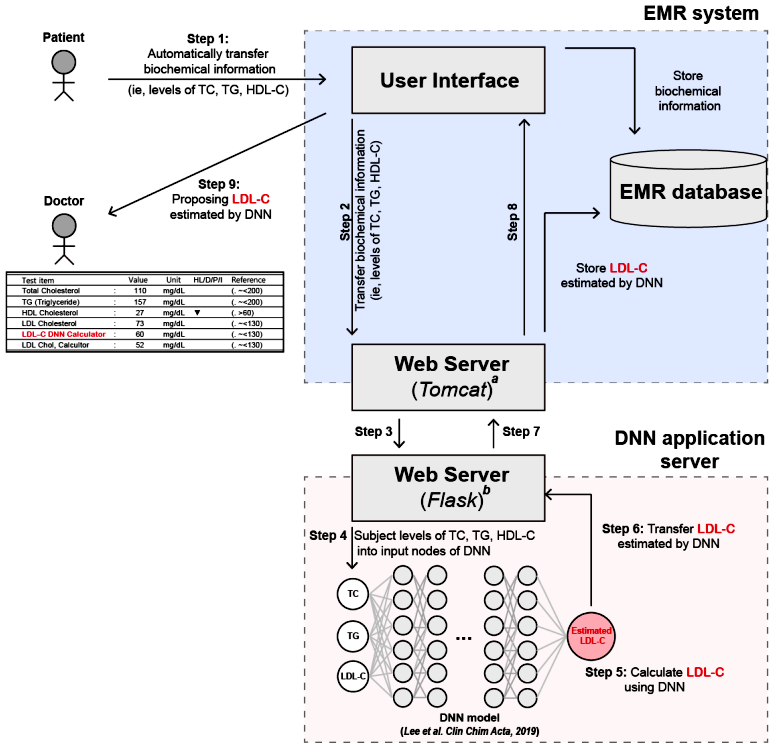
Fig 3[7]
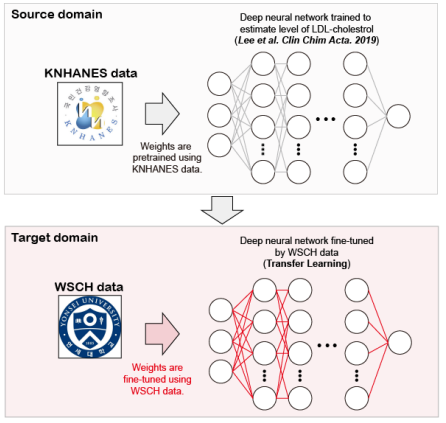
Fig 4[7]
인공지능, 기계학습, 그리고 딥러닝은 도구이면서 플랫폼이다. 상기 플랫폼을 적절하게 이용할 수 있다면 미래의 막대한 의료의 양을 감당 및 해석할 수 있을 것이다.
[참고문헌]
1. LeCun, Y.; Bengio, Y.; Hinton, G. Deep learning. Nature 2015, 521, 436-444.
2. Lee, T.; Lee, H. Prediction of Alzheimer's disease using blood gene expression data. Sci Rep 2020, 10, 3485, doi:10.1038/s41598-020-60595-1.
3. Friedewald, W.T.; Levy, R.I.; Fredrickson, D.S. Estimation of the concentration of low-density lipoprotein cholesterol in plasma, without use of the preparative ultracentrifuge. Clinical chemistry 1972, 18, 499-502.
4. Martin, S.S.; Blaha, M.J.; Elshazly, M.B.; Toth, P.P.; Kwiterovich, P.O.; Blumenthal, R.S.; Jones, S.R. Comparison of a novel method vs the Friedewald equation for estimating low-density lipoprotein cholesterol levels from the standard lipid profile. Jama 2013, 310, 2061-2068, doi:10.1001/jama.2013.280532.
5. Sampson, M.; Ling, C.; Sun, Q.; Harb, R.; Ashmaig, M.; Warnick, R.; Sethi, A.; Fleming, J.K.; Otvos, J.D.; Meeusen, J.W.; et al. A New Equation for Calculation of Low-Density Lipoprotein Cholesterol in Patients With Normolipidemia and/or Hypertriglyceridemia. JAMA Cardiol 2020, 5, 540-548, doi:10.1001/jamacardio.2020.0013.
6. Lee, T.; Kim, J.; Uh, Y.; Lee, H. Deep neural network for estimating low density lipoprotein cholesterol. Clin Chim Acta 2019, 489, 35-40, doi:10.1016/j.cca.2018.11.022.
7. Hwang, S.; Gwon, C.; Seo, D.M.; Cho, J.; Kim, J.Y.; Uh, Y. A Deep Neural Network for Estimating Low-Density Lipoprotein Cholesterol From Electronic Health Records: Real-Time Routine Clinical Application. JMIR Med Inform 2021, 9, e29331, doi:10.2196/29331.
